反硝化作用是在低氧或厌氧条件下, 由反硝化细菌介导以硝酸盐或亚硝酸盐作为末端电子受体还原成气态氮化合物(N2、NO和N2O)的生物异化过程[1], 该过程是生态系统氮循环的重要组成部分。具有反硝化功能的基因分布非常广泛, 细菌种群丰富, 与系统分化无关, 不能通过16S rRNA反映环境中反硝化细菌系统发育情况及群落结构特征[2], 利用反硝化功能基因研究不同土壤环境中的反硝化细菌群落成为目前的必然趋势。研究表明, 硝酸盐还原酶、亚硝酸盐还原酶、异化亚硝酸盐还原酶以及氧化亚氮还原酶是反硝化过程中4种互相独立的关键酶, 其中亚硝酸还原酶调控的是将亚硝酸盐还原成NO的反应, 该反应是区分硝酸盐呼吸菌和反硝化菌的标志性反应[3]。亚硝酸还原酶包含Cu-亚硝酸还原酶(nirK基因编码)和细胞色素cdl-亚硝酸还原酶(nirS基因编码)2种不同的结构形态, 其中nirS基因反硝化细菌以假单胞菌(Pseudomonas spp.)占优势; 而nirK基因存在于许多亲缘关系较远的菌株中[3]。因此, 通过研究反应环境中的nirK基因反硝化细菌的群落结构更具有真实性和代表性。
大量研究表明, nirK型反硝化细菌存在于农田、森林、草地、沉积物及水体等环境中[4-8], 并且发现环境性质的改变可以影响nirK型微生物的群落结构及多样性, 如NO3-、全氮含量、pH等[9], 同时微生物群落结构及多样性又能反映环境因子的综合作用。在我国南方稻作区, 水稻(Oryza sativa)种植与蛙、鱼、虾、鸭等水产经济动物养殖相结合形成的互利共生稻田种养复合生态模式是一种主要的种养模式[10]。水稻种植与克氏螯虾(Procambarus clarkii)共作形成的稻虾共作模式是稻田综合种养模式之一, 截至2016年, 仅在江汉平原推广面积已达20万hm2以上[11]。曹凑贵等[12]指出全国范围内适宜稻虾共作的面积占现有稻田总面积的15%, 具有较大的发展潜力。稻虾共作模式不仅具有极高的经济效益, 相比传统中稻模式, 该模式还可以改善土壤结构, 增加土壤养分[10]。但目前对于稻虾共作模式的生态研究多集中于宏观的N2O排放及土壤性质的研究。徐祥玉等[11]发现, 稻虾共作可以显著降低稻田N2O排放, 而稻田土壤N2O排放主要来自土壤微生物过程, 需要对稻虾共作土壤N2O排放的微生物调控机制进行深入研究。因此, 通过研究nirK基因型反硝化细菌的群落结构及多样性, 对于深入认识稻虾共作模式稻田反硝化作用的微生物调控机制具有十分重要的意义。
本文利用Illumina Miseq高通量测序技术对稻虾共作模式稻田土壤中nirK反硝化细菌的群落结构及多样性进行分析, 并通过冗余分析(RDA)来探索影响nirK反硝化细菌的关键因子, 以期揭示稻虾共作模式土壤反硝化细菌的多样性及群落结构的变化, 为深入认识该种植模式下反硝化作用微生物调控机制提供理论依据。
1 材料与方法 1.1 试验地概况与试验设计试验地位于湖北省荆州市长江大学试验基地(30°6′N, 111°54′E), 属江汉平原渍涝农田区域。该地区属亚热带季风气候, 为冲积性母质发育的水稻土, 年平均气温约16.4 ℃, 全年≥0 ℃积温6 228.4 ℃, 无霜期250 d, 年均降雨量1 148 mm, 年均日照时数2 000 h, 年太阳辐射总值约470 J·cm-2。
试验设置稻虾共作模式(CR)和常规中稻模式(MR)两个处理, 各处理分别设置3个小区, 随机区组设计, 小区面积为60 m2。水稻品种为‘丰两优香一号’。两种模式稻田均采用秸秆还田, 施肥管理采用当地常规措施。氮、磷、钾肥施用量分别为N 240 kg·hm-2、P2O5 120 kg·hm-2、K2O 120 kg·hm-2。氮肥按基肥:分蘖肥:拔节肥:穗肥=4:3:2:1施用, 磷肥一次性基施, 钾肥作基肥和长粗肥每次施用50%。两种模式水稻生长期间均采用前期灌水、中期晒田、后期干湿交替的水分管理模式。其他田间管理条件也相同。
稻虾共作模式稻田从2014年由常规中稻稻田改成。水稻移栽3 d后放养虾苗, 供试品种为‘克氏原鳌虾’, 投放密度为15.01~22.50万只·hm-2, 均匀分散投入虾沟, 冬季覆水, 虾沟上宽0.7 m, 下宽0.5 m, 深0.6 m, 虾沟围绕稻田四周, 呈闭合“口”字状。虾沟四周外围设尼龙拦虾网, 尼龙网沿虾沟外侧壁埋入地下约1 m, 地上高出稻田约0.3 m, 并用小竹桩支撑。稻田在晒田控蘖及落干时期, 虾随水迁移至虾沟中, 待复水后再次进入稻田生活。水稻生长期间不对克氏螯虾进行任何捕获, 在水稻收获前一天统一捕虾。常规中稻和稻虾共作模式均于每年5月中下旬进行整地、水稻移栽, 采用人工移栽, 宽行窄株, 株行距为16.7 cm×26.6 cm, 9月底收割, 稻草还田。
1.2 土壤样品采集于2016年水稻抽穗期(8月6日, 田间水层高度2~5 cm)取稻田表层(0~10 cm)土壤, 采用五点取样法, 采样点距离周围植株5~10 cm, 各小区采集5份土样均匀混合成1个样品, 除去根系、碎石及其他杂物后分为两部分。一部分迅速用灭菌锡箔纸包裹, 放入液氮罐中低温保存, 带回实验室后放入超低温冰箱, -80 ℃保存, 用于微生物研究; 另一部分鲜土采用自封袋密封后带回实验室放入冰箱, -4 ℃保存, 测定铵态氮和硝态氮, 该部分剩余土风干后过100目筛测定土壤pH、碱解氮、全碳和全氮含量。
1.3 土壤理化性质测定土壤pH采用电位法(水:土=2.5:1)测定; 土壤全碳、全氮均使用元素分析仪(ECS4024, Costech, Italy)测定[13]; 土壤铵态氮用1 mol·L-1 KCl溶液浸提新鲜土样后, 在200 r·min-1下振荡浸提1 h, 静置, 过滤, 采用靛酚蓝比色法测定[14]; 硝态氮采用203 nm和230 nm双波长紫外分光光度法测定[15]; 土壤碱解氮采用碱解扩散法测定。
1.4 DNA提取及PCR扩增取0.25 g新鲜土壤样品, 采用PowerSoil®DNA Isolation Kit(Mobio, USA)试剂盒[16]提取土壤DNA。完成基因组DNA抽提后, 利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。
选用引物nirK1aCu(5'-ATCATGGTSCTGCCG-CG-3')和nirKR3Cu(5'-GCCTCGATCAGRTTGTGG-TT-3')扩增nirK基因[17]。PCR采用TransStart Fastpfu DNA Polymerase(TransGen AP221-02, 北京, 中国)在PCR仪ABI GeneAmp® 9 700(ABI, CA, USA)上进行。每个样本3个重复, 将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测, 使用AxyPrepDNA凝胶回收试剂盒(AXYGEN, CA, USA)切胶回收PCR产物, Tris_HCl洗脱; 2%琼脂糖电泳检测。PCR反应体系为20 μL, 包括: 2×sybr MIX (with ROX)10 μL, 两种上、下游引物(10 μmol·L-1)各0.2 μL, 样本1 μL(稀释8倍), 用RNase-Free ddH2O补足至20 μL。程序设置: 95 ℃预变性600 s; 94 ℃ 20 s, 58.6 ℃ 20 s, 72 ℃ 40 s, 共40个循环; 72 ℃10 min。
将PCR产物回收, 连接至pUC-T载体(CWBIO, 北京, 中国)上, 转化大肠埃希菌DH5a后进行培养, 选择阳性克隆菌株提取质粒, 采用核酸检测仪检测浓度和纯度, 将浓度换算成拷贝数, 梯度稀释, -80 ℃保存, 用于制备标准曲线。
1.5 荧光定量、Illumina Miseq测序及序列筛选参照电泳初步定量结果, 将PCR产物用QuantiFluor™-ST蓝色荧光定量系统(Promega公司)进行检测定量, 之后按照每个样本的测序量要求, 进行相应比例的混合, 然后用Illmumina Miseq测序平台进行双末端测序, 测序服务委托北京美吉桑格生物医药科技有限公司完成。
MiSeq测序得到的是双端序列数据, 使用FLASH和Trimmomatic软件首先根据PE reads之间的overlap关系, 将成对的reads拼接(merge)成1条序列, 同时对reads的质量和merge的效果进行质控过滤, 根据序列首尾两端的barcode和引物序列区分样品得到有效序列, 并校正序列方向, 即为优化数据, 数据去杂方法和具体参数设置依据杨亚东等[18]研究。
1.6 数据处理使用Usearch 7.1软件将优质序列聚类成操作分类单元(Operational Taxonomic Units, OTU), 按照97%相似性对重复序列(不含单序列)进行OTU聚类, 采用RDP classifier分类法对97%相似水平的OTU代表序列进行分类学分析; 使用Mothur 1.30.1软件在相似水平97%上进行微生物多样性指数评估。数据方差分析和相关性分析用SPSS 20.0软件完成, 采用单因素方差分析法区分样品间的显著性差异(n=3, Tukey, P < 0.05);利用R语言工具制作曲线图及Pearson热图, 利用CANOCO 5.0软件对土壤理化性质和nirK基因反硝化群落结构进行冗余分析(RDA)。
2 结果与分析 2.1 稻虾共作对土壤相关理化性质的影响在0~10 cm土层土壤中, 稻虾共作模式(CR)土壤硝态氮、全碳、全氮含量均显著高于常规中稻模式(MR)(P < 0.05)(表 1)。pH CR低于MR, 差异不显著(7.41~7.47, P > 0.05);碱解氮和铵态氮含量CR均高于MR, 差异不显著(P > 0.05);碳氮比CR低于MR, 差异不显著。研究结果表明, 稻虾共作模式可显著提高水稻抽穗期稻田土壤中硝态氮、全氮及全碳的含量, 对碳氮比和碱解氮、铵态氮含量没有显著影响。
| 表1 稻虾共作模式对稻田土壤理化性质的影响 Table 1 Effects of integrated rice-crayfish farming on soil physicochemical properties in paddy field |
采用Miseq sequencing技术对微生物nirK基因测序分析, 数据经过优化筛选后6个样品共测得原始序列209 777条, 序列平均长度为448.16 bp, 共测得碱基94 009 020个, 所有样品的序列长度在202~537 bp(表 2)。按97%的相似度对非重复序列进行OTU分析, 共得到344个OTUs。对各样本序列进行随机抽样的方法, 以抽到的序列数与它们对应的物种多样性指数, 构建稀释曲线(rarefaction curves)。样本Coverage指数在0.996 6~0.997 8, 稀释性曲线均趋于平坦饱和, 表明此测序深度获得序列数据量可以反映土壤样品nirK基因微生物信息(图 1)。
| 表2 稻虾共作模式和常规中稻模式3次重复土壤样品测序结果 Table 2 Sequencing results of three repeated soil samples from integrated rice-crayfish farming system and conventional middle rice system |

|
图 1 稻虾共作模式(CR)和常规中稻模式(MR)土壤样品OTU水平Coverage指数稀释性曲线 Figure 1 Rarefaction curves of OTU level coverage indexes in soil samples of integrated rice-crayfish farming system (CR) and conventional middle rice system (MR) |
对Alpha多样性指数进行单因素方差分析(表 3), 结果显示两处理间Shannon指数和Simpson指数差异不显著, CR处理的Sobs指数、Ace指数和Chao指数均显著高于MR处理。Sobs指数、Ace指数、Chao指数是衡量微生物群落丰富度指数, Shannon指数和Simpson指数是衡量微生物群落多样性的重要指标。结果表明, 稻虾共作模式(CR)较常规中稻模式(MR)显著增加了稻田土壤nirK基因微生物的群落丰富度, 未显著改变nirK基因微生物群落的多样性。
| 表3 稻虾共作模式(CR)和常规中稻模式(MR)Alpha多样性指数 Table 3 Alpha diversity indices of integrated rice-crayfish farming system (CR) and conventional middle rice system (MR) |
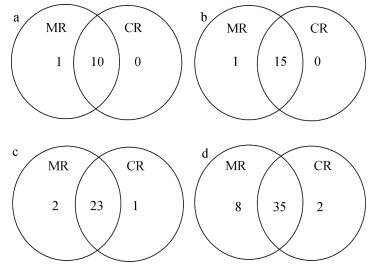
在水稻抽穗期, 对CR处理和MR处理nirK基因微生物进行目、科、属、种的物种分类学水平分析(图 2)。在目水平上, CR处理和MR处理具有相同的10个目(图 2a), MR处理独有红螺菌目(Rhodospirillales)。在科水平上, CR处理和MR处理具有相同的15个科(图 2b), MR处理独有红螺菌科(Rhodospirillaceae)。在属水平上, CR处理和MR处理具有相同的23个属(图 2c), MR处理独有2个属, 分别是红螺菌目红螺菌科的固氮螺菌属(Azospirillum)和根瘤菌目根瘤菌科的中华根瘤菌属(Sinorhizobium), CR处理独有盐杆菌目盐杆菌科的盐惰菌属(Halopiger)。在种水平上, CR处理和MR处理具有相同的35个种(图 2d), MR独有8个种, 分别是红假单胞菌属的Rhodopseudomonas_sp._2-8、Rhodopseudomonas_palustris, 中华根瘤菌属的Sinorhizobium_sp._R-24605, 根瘤菌属的Rhizobium_ sp._R-24658, 包西氏菌属的Bosea_sp._MF18, 中慢生根瘤菌属的Mesorhizobium_sp._D237c, norank_p__environmental_samples_k__norank属的uncultured_ marine_bacterium, 固氮螺菌属的Azospirillum_sp._ TSH20; CR独有2个种, 分别是norank_p__environmental_samples_k__norank属的uncultured_bacterium_d__Archaea和盐惰菌属的Halopiger_xanaduensis。分析表明, 稻虾共作模式改变了常规稻田模式的nirK基因微生物在目、科、属、种水平的群落组成, 较常规中稻模式, 稻虾共作模式在各分类水平组成类群均减少。
|
图 2 稻虾共作模式(CR)和常规中稻模式(MR)土壤样品nirK基因微生物物种分类学组成Venn图 Figure 2 Venn drawings of nirK gene species taxonomy of integrated rice-crayfish farming system (CR) and conventional middle rice system (MR) a、b、c、d分别表示目、科、属、种4种分类学水平。 a, b, c and d respectively show the taxonomic levels of order, family, genus and species. |
两处理所有土壤样品获得的nirK基因微生物物种分类在3个界、5个门、7个纲、11个目、16个科、26个属和45个种。将无法分类的序列定义为无法归类。两处理所有样品获得的nirK基因OTUs在分类学界、门、纲、目、科、属、种水平上可归类比例为97.8%、96.0%、93.0%、89.0%、84.4%、73.3%和60.0%。
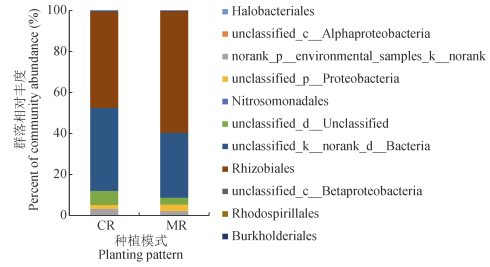
在目水平上, Rhizobiales(根瘤菌目)、unclassified_k__norank_d__Bacteria、unclassified_d__Unclassified、unclassified_p__Proteobacteria和norank_p__environmental_samples_k__norank为两处理平均相对丰度同时大于1%的5个优势目(图 3), CR处理3次重复平均相对丰度分别为47.0%、40.5%、6.9%、2.0%和3.0%, MR处理3次重复平均相对丰度分别为59.4%、31.7%、3.3%、3.2%和2.0%, 两处理中的优势目平均相对丰度差异没有达到显著水平(P > 0.05)。Burkholderiales (伯克氏菌目)、Rhodospirillales(红螺菌目)、unclassified_c__Betaproteobacteria为MR处理平均相对丰度介于0.01%~1%的目, 平均相对丰度分别为0.18%、0.07%、0.06%, 在CR处理中平均相对丰度分别为0.19%、0、0.35%, 其中Burkholderiales和unclassified_c__Betaproteobacteria在两处理中相对丰度没有达到显著性差异, CR处理缺失Rhodospirillales (红螺菌目)。两处理共有目的相对丰度差异不显著, 稻虾共作模式较常规中稻模式改变了目的种类, 对共有目相对丰度没有显著性改变。
|
图 3 稻虾共作模式(CR)和常规中稻模式(MR)样品nirK基因目水平组成 Figure 3 Order distributions of nirK gene in different soil samples of integrated rice-crayfish farming system (CR) and conventional middle rice system (MR) Halobacteriales:盐细菌; Burkholderiales:伯克霍尔德氏菌; unclassified_c_Betaproteobacteria:未分类c-β蛋白酶细菌; unclassified_p_proteobacteria:未分类p-蛋白细菌; Rhizobiales:根瘤菌; unclassified_c__Alphaproteobacteria:未分类_c__α-蛋白细菌; Nitrosomonadales:亚硝化单胞菌目; Rhodospirillales:红螺菌目。 |
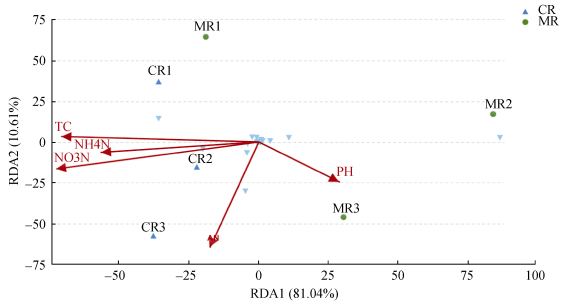
对97%相似水平的OTU代表序列进行DCA分析, 如果Lengths of gradient第1轴的值大于4.0, 选CCA分析; 如果介于3.0~4.0, 选RDA和CCA分析均可; 如果小于3.0, RDA分析优于CCA分析。分析结果中Lengths of gradient第1轴的大小为0.906, 选用RDA分析环境因子、样本、菌群三者间的关系。
RDA分析的前两个排序轴共解释了91.65%的群落变化(图 4), 第1排序轴(RDA1)解释了81.04%, 第2排序轴(RDA2)解释了10.61%。第1排序轴与土壤总碳(TC)、铵态氮(NH4+-N)、硝态氮(NO3--N)关系最为密切; 第2排序轴与碱解氮(AN)关系最为密切。总体而言, 土壤硝态氮(P=0.019)对细菌群落的影响最显著。沿第1轴看, CR1、CR2、CR3和MR1样本距离较近, 趋于聚集在一起, 表明这4个样本的群落结构相近; 相比之下, MR2与MR3样本距离较近, 表明样本群落结构相近, MR2和MR3与MR1距离较远, 说明空间异质性对微生物群落结构存在影响。同时, MR2、MR3样本与CR1、CR2和CR3距离较远, 表明这两组样本群落结构存在差异, 综合表明稻虾共作模式对土壤nirK基因微生物的群落结构有一定的改变, 但稻虾模式与常规中稻模式在群落结构上仍保留着一定的相似性, 硝态氮含量是影响nirK反硝化细菌群落结构的主效因子。
|
图 4 稻虾共作模式和常规中稻模式土壤化学性质与细菌群落(属水平)的冗余分析 Figure 4 Results from redundancy analysis to explore the relationship between soil bacterial community (at genus level) and soil physiochemical characteristics of integrated rice-crayfish farming system and conventional mid-rice model MR1、MR2和MR3分别表示常规中稻模式的3次重复, CR1、CR2和CR3分别表示稻虾共作模式的3次重复。TC:土壤总碳; NO3N:硝态氮; NH4N:铵态氮; PH: pH; AN:碱解氮。 MR1, MR2 and MR3 mean three replicates of the conventional middle rice; CR1, CR2 and CR3 mean three replicates of the rice-crayfish culture. TC: soil total carbon; NO3N: nitrate nitrogen; NH4N: ammonium nitrogen; PH: pH; AN: available nitrogen. |
Delmont等[19]研究表明, 土壤微生物群落结构及多样性会随着土壤理化性质的改变而变化, 土壤理化性质在一定程度上决定着土壤微生物结构。克氏螯虾在稻田取食、蜕皮和排泄等活动, 可能会增加土壤中营养物质, 导致土壤全氮和全碳含量增加[12]。本研究表明, 稻虾模式较常规中稻模式稻田土壤全氮、全碳含量显著增加, 结果与前人研究结果一致[20]。稻虾共作模式由于克氏螯虾营底栖生活, 其爬行、潜穴等活动会导致土壤通透性增加[21], 有研究表明, 穴居活动会显著影响土壤与环境之间的水、气等物质交换[22-23], 在通气良好的情况下部分铵态氮会转化成硝态氮, 可能会导致稻虾模式土壤硝态氮含量增加。本研究中, 相比常规中稻模式, 稻虾土壤中全氮、硝态氮及全氮含量显著提升, nirK微生物丰富度显著增加, 结果与Chen等[24]在研究中得出土壤中全氮、硝态氮及有机质的改变会引起土壤中微生物群落的变化结论相一致。克氏螯虾等底栖动物通过爬行、觅食和潜穴等生物扰动改变了营养物质及氧气的供应, 可能影响着微生物群落结构及丰富度[25]。本研究中nirK功能基因微生物多样性指数没有显著改变可能与稻虾种植年限有关, 3年内环境因子变化可能对稻虾表层土壤(0~10 cm)nirK功能基因微生物多样性的影响还是有限的。
稻虾共作模式与常规中稻模式的微生物群落结构组成在目、科、属、种水平上存在差异。相比MR处理, 稻虾共作模式缺失红螺菌目、红螺菌科、Azospirillum(固氮螺菌属)及根瘤菌目根瘤菌科的Sinorhizobium(中华根瘤菌属), 稻虾共作模式增加盐杆菌目盐杆菌科的Halopiger(盐惰菌属)。稻虾共作模式缺失的红螺菌目和根瘤菌目均具有固氮和固碳的功能[25]。梅志平等[26]研究表明红螺菌具有一定的饥饿存活能力, 同时红螺菌在缺失有机物的生长环境下会利用光能生产有机物, 在有可利用的有机物条件下, 便自行生长[27], 因此稻虾共作模式缺失红螺菌不是因为缺乏红螺菌生存所需有机物。尤希凤等[28]研究表明在低于2 000 lx光照条件下, 红螺菌随着光照强度的增加生长速度加快, 同时红螺菌是典型的厌氧细菌, 氧气的存在制约着红螺菌的生长。一方面, 采样期水稻株高120~130 cm, 生长旺盛, 田间土壤光照较弱不利于土壤中红螺菌的生长; 另一方面, 田间水层高度2~5 cm, 稻虾共作模式中克氏螯虾的扰动可能会增加土壤中的氧气, 抑制红螺菌的生长。胡千德等[29]研究表明中华根瘤菌属的模式种费氏中华根瘤菌(Sinorhizobium fredii)的耐盐性较弱, 本研究中稻虾共作模式的土壤硝酸盐含量显著高于常规中稻模式, 可能是导致稻虾共作模式缺失中华根瘤菌的原因之一。Halopiger(盐惰菌属)是稻虾共作模式后增加的菌属, 目前国内外关于稻虾养殖中增加盐惰菌属原因的研究较少, 具体原因有待进一步深入研究。克氏螯虾的生命活动可能导致稻田土微域生境条件如含氧量、氧化还原电位等发生改变[10], 微域生境条件综合性质的改变可能是导致稻虾模式微生物群落结构发生变化的直接原因。前人研究表明, 红螺菌能大幅降低水体有机质含量、增加溶氧、改善水质[30], 对Cu2+、Pb2+等重金属离子有较强的吸附作用[31-32]。本研究表明稻虾共作模式会导致土壤中缺失红螺菌, 因此, 在稻虾共作实际生产中增施红螺菌有助于提升水体净化能力, 净化水质, 有助于虾的生长, 增加产量。
环境因子与反硝化细菌群落的冗余分析(RDA)发现硝态氮含量是影响nirK反硝化细菌群落结构的主效因子。Xie等[33]通过研究青藏高原草甸土放牧对反硝化细菌群落结构的影响, 发现硝态氮是影响nirK反硝化细菌群落结构的重要因子, 与本文结论相一致。硝态氮作为反硝化反应的起始底物之一, 是控制nirK型反硝化细菌进行反硝化作用的重要因素。冗余分析表明, CR处理与MR处理微生物群落结构既有差异性, 又保持一定的相似性, 该结果与群落物种组成分析和群落目水平相对丰度分析结果一致。
4 结论与常规中稻模式相比, 连续3年稻虾共作模式可提高稻田土壤全氮、全碳、硝态氮含量, 对稻田土壤nirK基因微生物的多样性指数无显著影响, 但显著增加了稻田土壤nirK基因微生物群落丰富度指数; 较常规中稻模式, 稻虾共作模式没有显著改变nirK基因共有物种目水平的相对丰度, 但改变了稻田土壤中微生物nirK基因在目、科、属、种水平的群落组成, 硝态氮含量是影响nirK反硝化细菌群落结构的主效因子。稻虾共作模式会导致土壤中缺失红螺菌, 因此, 在稻虾共作实际生产中适当增施红螺菌有助于提高水体净化能力, 改善虾生长水体环境, 有利于克氏螯虾的生长, 增加稻虾经济收入。
| [1] |
BARDON C, POLY F, PIOLA F, et al. Mechanism of biological denitrification inhibition:Procyanidins induce an allosteric transition of the membrane-bound nitrate reductase through membrane alteration[J]. FEMS Microbiology Ecology, 2016, 92(5): Fiw034. DOI:10.1093/femsec/fiw034 |
| [2] |
ZUMFT W G. Cell biology and molecular basis of denitrification[J]. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533-616. |
| [3] |
BRAKER G, ZHOU J Z, WU L Y, et al. Nitrite reductase genes (nirK and nirS) as functional markers to investigate diversity of denitrifying bacteria in pacific northwest marine sediment communities[J]. Applied and Environmental Microbiology, 2000, 66(5): 2096-2104. DOI:10.1128/AEM.66.5.2096-2104.2000 |
| [4] |
YANG Y D, ZHAO J, JIANG Y, et al. Response of bacteria harboring nirS and nirK genes to different N fertilization rates in an alkaline northern Chinese soil[J]. European Journal of Soil Biology, 2017, 82: 1-9. DOI:10.1016/j.ejsobi.2017.05.006 |
| [5] |
JHA N, SAGGAR S, GILTRAP D, et al. Soil properties impacting denitrifier community size, structure, and activity in New Zealand dairy-grazed pasture[J]. Biogeosciences Discussions, 2016, 14(18): 1-19. |
| [6] |
CHEN J, NIE Y X, LIU W, et al. Ammonia-oxidizing archaea are more resistant than denitrifiers to seasonal precipitation changes in an acidic subtropical forest soil[J]. Frontiers in Microbiology, 2017, 8: 1384. DOI:10.3389/fmicb.2017.01384 |
| [7] |
PARK S, KIM H K, KIM M S, et al. Monitoring nitrate natural attenuation and analysis of indigenous micro-organism community in groundwater[J]. Desalination and Water Treatment, 2016, 57(51): 24096-24108. DOI:10.1080/19443994.2016.1145955 |
| [8] |
JIANG X L, YAO L, GUO L D, et al. Multi-scale factors affecting composition, diversity, and abundance of sediment denitrifying microorganisms in Yangtze lakes[J]. Applied Microbiology and Biotechnology, 2017, 101(21): 8015-8027. DOI:10.1007/s00253-017-8537-5 |
| [9] |
HAN B, YE X H, LI W, et al. The effects of different irrigation regimes on nitrous oxide emissions and influencing factors in greenhouse tomato fields[J]. Journal of Soils and Sediments, 2017, 17(10): 2457-2468. DOI:10.1007/s11368-017-1700-x |
| [10] |
佀国涵, 彭成林, 徐祥玉, 等. 稻虾共作模式对涝渍稻田土壤理化性状的影响[J]. 中国生态农业学报, 2017, 25(1): 61-68. SI G H, PENG C L, XU X Y, et al. Effect of integrated rice-crayfish farming system on soil physico-chemical properties in waterlogged paddy soils[J]. Chinese Journal of Eco-Agriculture, 2017, 25(1): 61-68. |
| [11] |
徐祥玉, 张敏敏, 彭成林, 等. 稻虾共作对秸秆还田后稻田温室气体排放的影响[J]. 中国生态农业学报, 2017, 25(11): 1591-1603. XU X Y, ZHANG M M, PENG C L, et al. Effect of rice-crayfish co-culture on greenhouse gases emission in straw-puddled paddy fields[J]. Chinese Journal of Eco-Agriculture, 2017, 25(11): 1591-1603. |
| [12] |
曹凑贵, 江洋, 汪金平, 等. 稻虾共作模式的"双刃性"及可持续发展策略[J]. 中国生态农业学报, 2017, 25(9): 1245-1253. CAO C G, JIANG Y, WANG J P, et al. "Dual character" of rice-crayfish culture and strategies for its sustainable development[J]. Chinese Journal of Eco-Agriculture, 2017, 25(9): 1245-1253. |
| [13] |
王恒生, 刁治民, 陈克龙, 等. 青海湖流域小泊湖湿地土壤微生物数量及影响因子[J]. 中国农业大学学报, 2015, 20(6): 189-197. WANG H S, DIAO Z M, CHEN K L, et al. Quantity of soil microbe and affecting factors of Xiaopohu Wetlands in Qinghai Lake Basin[J]. Journal of China Agricultural University, 2015, 20(6): 189-197. |
| [14] |
詹晓燕, 刘臣辉, 范海燕, 等. 水体中氨氮测定方法的比较——纳氏试剂光度法、靛酚蓝比色法[J]. 环境科学与管理, 2010, 35(11): 132-134. ZHAN X Y, LIU C H, FAN H Y, et al. Comparison between two N-ammoniacal measurements in water-Napierian reagent colorimetric method and indophenol-blue colorimetric method[J]. Environmental Science and Management, 2010, 35(11): 132-134. DOI:10.3969/j.issn.1673-1212.2010.11.034 |
| [15] |
涂常青, 温欣荣. 双波长分光光度法测定土壤硝态氮[J]. 中国土壤与肥料, 2006(1): 50-51. TU C Q, WEN X R. Determination of nitric nitrogen in soil extracts by dual wavelength spectrophotometric method[J]. Soil and Fertilizer Sciences in China, 2006(1): 50-51. DOI:10.3969/j.issn.1673-6257.2006.01.013 |
| [16] |
王国兴, 董桂军, 艾士奇, 等. 通风量对堆肥化过程中氮素转化及nirK基因多样性和数量的影响[J]. 农业环境科学学报, 2016, 35(3): 565-572. WANG G X, DONG G J, AI S Q, et al. Effects of ventilation on nitrogen transformation and nirK gene diversity and abundance during comp[J]. Journal of Agro-Environment Science, 2016, 35(3): 565-572. |
| [17] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624. DOI:10.1038/ismej.2012.8 |
| [18] |
杨亚东, 冯晓敏, 胡跃高, 等. 豆科作物间作燕麦对土壤固氮微生物丰度和群落结构的影响[J]. 应用生态学报, 2017, 28(3): 957-965. YANG Y D, FENG X M, HU Y G, et al. Effects of legume-oat intercropping on abundance and community structure of soil N2-fixing bacteria[J]. Chinese Journal of Applied Ecology, 2017, 28(3): 957-965. |
| [19] |
DELMONT T O, FRANCIOLI D, JACQUESSON S, et al. Microbial community development and unseen diversity recovery in inoculated sterile soil[J]. Biology and Fertility of Soils, 2014, 50(7): 1069-1076. DOI:10.1007/s00374-014-0925-8 |
| [20] |
SI G H, PENG C L, YUAN J F, et al. Changes in soil microbial community composition and organic carbon fractions in an integrated rice-crayfish farming system in subtropical China[J]. Scientific Reports, 2017, 7: 2856. DOI:10.1038/s41598-017-02984-7 |
| [21] |
WANG J Q, ZHANG X D, LI F, et al. Bioturbation of burrowing crabs promotes sediment turnover and carbon and nitrogen movements in an estuarine salt marsh[J]. Ecosystems, 2010, 13(4): 586-599. DOI:10.1007/s10021-010-9342-5 |
| [22] |
DALEO P, IRIBARNE O. The burrowing crab Neohelice granulata affects the root strategies of the cordgrass Spartina densiflora in SW Atlantic salt marshes[J]. Journal of Experimental Marine Biology and Ecology, 2009, 373(1): 66-71. DOI:10.1016/j.jembe.2009.03.005 |
| [23] |
WEISSBERGER E J, COIRO L L, DAVEY E W. Effects of hypoxia on animal burrow construction and consequent effects on sediment redox profiles[J]. Journal of Experimental Marine Biology and Ecology, 2009, 371(1): 60-67. DOI:10.1016/j.jembe.2009.01.005 |
| [24] |
CHEN C, ZHANG J N, LU M, et al. Microbial communities of an arable soil treated for 8 years with organic and inorganic fertilizers[J]. Biology and Fertility of Soils, 2016, 52(4): 455-467. DOI:10.1007/s00374-016-1089-5 |
| [25] |
HESSELSOE M, FÜREDER S, SCHLOTER M, et al. Isotope array analysis of Rhodocyclales uncovers functional redundancy and versatility in an activated sludge[J]. The ISME Journal, 2009, 3(12): 1349-1364. DOI:10.1038/ismej.2009.78 |
| [26] |
梅志平, 章宗涉. 红螺菌(Rhodospirillum sp.)的生长及其饥饿存活的研究[J]. 生态学报, 2000, 20(1): 118-123. MEI Z P, ZHANG Z S. Studies on the growth and starvation-survival of Rhodospirillum sp[J]. Acta Ecologica Sinica, 2000, 20(1): 118-123. |
| [27] |
刘璐. 有益微生物在水产养殖中的应用[J]. 江西农业, 2017(2): 106-107. LIU L. Application of beneficial microorganisms in aquaculture[J]. Jiangxi Agriculture, 2017(2): 106-107. |
| [28] |
尤希凤, 张全国, 杨群发, 等. 天然混合产氢红螺菌培养条件[J]. 太阳能学报, 2006, 27(4): 331-334. YOU X F, ZHANG Q G, YANG Q F, et al. The best culture conditions of natural mixture hydrogen producing rhodobacter sphaeroides[J]. Acta Energiae Solaris Sinica, 2006, 27(4): 331-334. DOI:10.3321/j.issn:0254-0096.2006.04.003 |
| [29] |
胡千德, 周俊初. 湖北潜江灰潮土中费氏中华根瘤菌(Sinorhizobium fredii)多样性的研究[J]. 微生物学杂志, 2000, 20(2): 7-10. HU Q D, ZHOU J C. Biodiversity of Sinorhizobium fredii in gray soil, Qianjiang, Hubei[J]. Journal of Microbiology, 2000, 20(2): 7-10. |
| [30] |
王兰, 廖丽华. 光合细菌的分离鉴定及对养殖水的净化研究[J]. 微生物学杂志, 2004, 24(2): 7-9. WANG L, LIAO L H. Separation and identification of photosynthetic bacteria (PSB) and purification of aquiculture water[J]. Journal of Microbiology, 2004, 24(2): 7-9. DOI:10.3969/j.issn.1005-7021.2004.02.003 |
| [31] |
黄富荣, 尹华, 彭辉, 等. 红螺菌对Cu2+的吸附研究[J]. 工业微生物, 2005, 35(1): 16-20. HUANG F R, YIN H, PENG H, et al. Studies on copper biosorption by Rhodospirillum[J]. Industrial Microbiology, 2005, 35(1): 16-20. DOI:10.3969/j.issn.1001-6678.2005.01.004 |
| [32] |
刘大岭, 林伟雄, 梁郁强. 红螺菌对铅离子吸附作用的初步研究[J]. 粮食与饲料工业, 2005(4): 38-40. LIU D L, LIN W X, LIANG Y Q. Preliminary study on sorption of lead ion by Rhodospirillum[J]. Cereal & Feed Industry, 2005(4): 38-40. DOI:10.3969/j.issn.1003-6202.2005.04.017 |
| [33] |
XIE Z, LE ROUX X, WANG C P, et al. Identifying response groups of soil nitrifiers and denitrifiers to grazing and associated soil environmental drivers in Tibetan alpine meadows[J]. Soil Biology and Biochemistry, 2014, 77: 89-99. DOI:10.1016/j.soilbio.2014.06.024 |
 2018, Vol. 26
2018, Vol. 26



